모델 설명
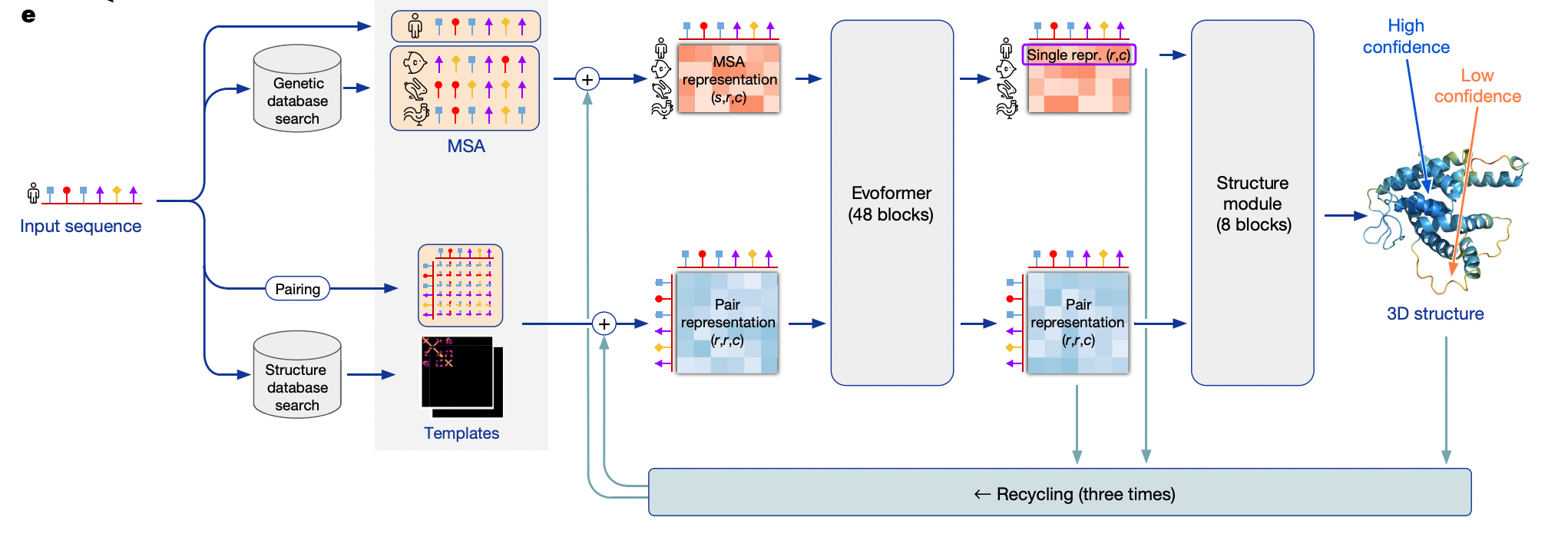
알파폴드의 입력으로 단백질의 아미노산 시퀀스가 들어간다.
- genetic database search를 통해서 MSA 를 구하고, MSA representation으로 나타낸다.
- 아미노산에대해서 pairing을 하고, structure database search를 통해서 template을 넣는다. pair representation으로 나타낸다.
<입력 부분>
이 부분에 대해서 조금 자세히 보도록 하자.
supplementary Figure 1이다.
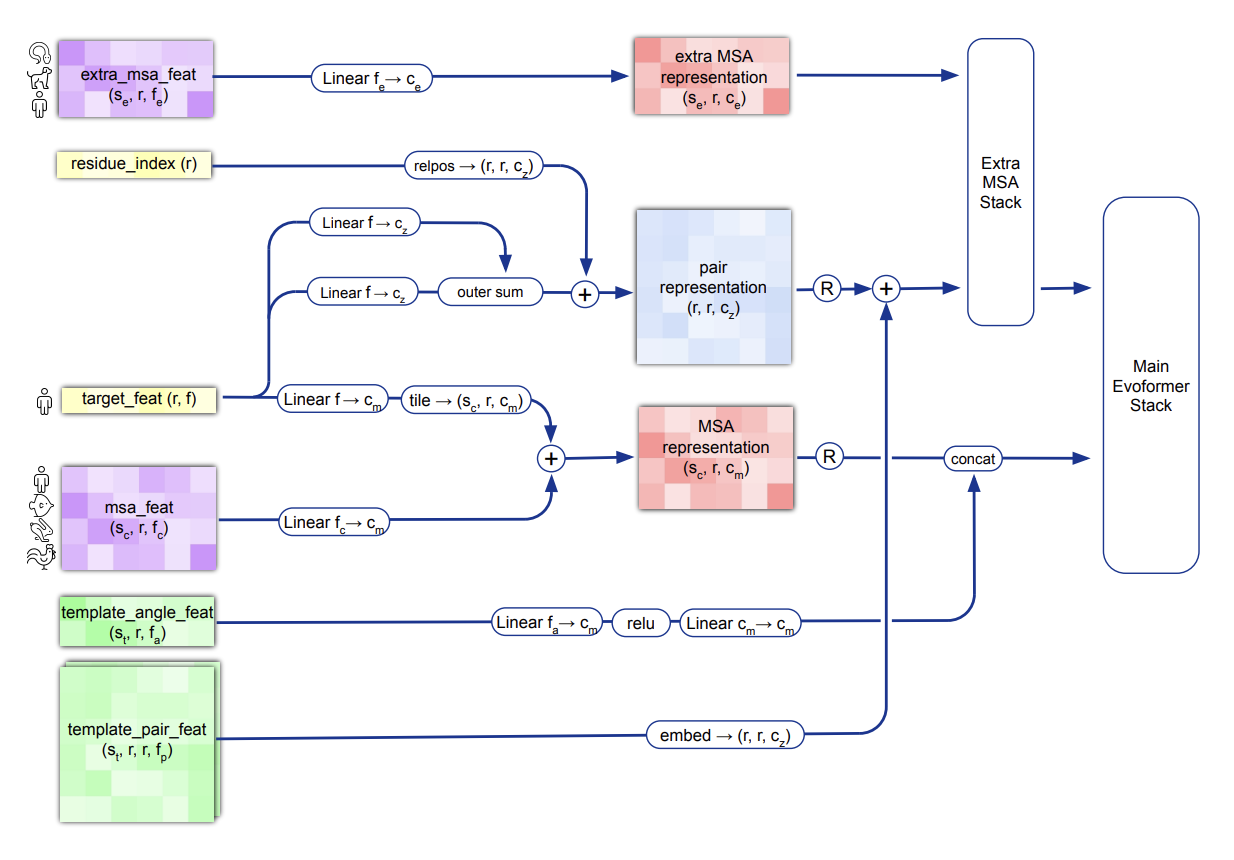
-맨 위에 보이는 extra msa feat은 옵션이다. 넣어줘도 되고 안넣어줘도 된다.
-pair representation 만들기 : residue_index에 대해서 (r,r,cz) 차원으로 변환을 시켜준다. target feat에 여러 linear layer를 거쳐서 차원을 변환시켜준 후 outer sum을 하고, 변환된 residue_index에 대해 더해주어서 pair representation을 만들어준다. 그리고 template_pair_feat을 더해준다.( 만약, template이 없는 단백질 시퀀스에 대해서는 template_pair_feat을 안더해줄것이다)
-> pair representation은 단백질 아미노산의 공간적인 특징을 가진다.
-MSA representation 만들기 : target_feat을 linear layer를 통해 차원을 변환시켜준 후, sc개 만큼 쌓아준다(tile). 그러면 (r,cm) 차원이 (sc,r,cm)차원으로 변환될 것이다. msa_feat을 linear layer를 통해 차원을 변환시켜주고, target_feat, msa_feat을 더해서 MSA representation을 만들어준다. template angle feature를 차원에 더해준다.
->MSA representation은 단백질 아미노산의 진화적인 특징을 가진다.
이렇게해서 pair representation과 MSA representation이 Evoformer의 입력값으로 들어간다.
'연구' 카테고리의 다른 글
| Alphafold2 논문 리뷰4 [Evoformer - pair representation] (0) | 2022.10.25 |
|---|---|
| Alphafold2 논문 리뷰3 [Evoformer - MSA representation] (0) | 2022.10.25 |
| Alphafold2 논문 리뷰1 - 성능 (1) | 2022.10.25 |
| SSPro 사용해서 단백질 2차구조 예측하기 (1) | 2022.09.22 |
| margianl distribution (주변 분포) (0) | 2022.09.20 |