반응형
dgl (deep graph library) 를 이용해서 SMILES를 그래프로 표현하는 방법을 알아보겠다!
아스피린을 예로 들어보겠다.
아스피린의 SMILES는 CC(=O)OC1=CC=CC=C1C(=O)O 이다. (Pubchem에서 가져옴)
그리고 그림을 그려보면,
from rdkit.Chem.Draw import rdMolDraw2D
from IPython.display import SVG
smi='CC(=O)OC1=CC=CC=C1C(=O)O'
mol = Chem.MolFromSmiles(smi)
이렇게 생겼다!!
이제 그래프로 바꿔보겠다.
from rdkit import Chem
from rdkit.Chem import MolFromSmiles
from dgllife.utils import BaseBondFeaturizer, bond_type_one_hot,bond_is_conjugated_one_hot,bond_is_in_ring_one_hot,bond_stereo_one_hot
from dgllife.utils import ConcatFeaturizer,BaseAtomFeaturizer, atom_type_one_hot, atom_degree_one_hot, atom_is_aromatic_one_hot
from dgllife.utils import smiles_to_bigraph
import dgl
smi='CC(=O)OC1=CC=CC=C1C(=O)O'
#atom feature
atom_featurizer = BaseAtomFeaturizer(featurizer_funcs = {'h': ConcatFeaturizer(
[atom_type_one_hot,atom_degree_one_hot,atom_is_aromatic_one_hot] )})
#bond feature
bond_featurizer = BaseBondFeaturizer({'e':ConcatFeaturizer([bond_type_one_hot,bond_is_conjugated_one_hot,bond_is_in_ring_one_hot,bond_stereo_one_hot])})
dgl.add_self_loop(smiles_to_bigraph(smi, node_featurizer=atom_featurizer, edge_featurizer = bond_featurizer))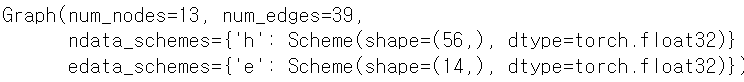
그러면 노드(atom)의 갯수가 13개, edge(bond)의 갯수가 39개인 graph가 만들어진다.
ndata는 node의 feature이다. atom featurizer에 atom type, atom degree, aromatic을 넣어주었다.
edata는 bond의 feature이고, bond featurizer에 bond type, conjugate, ring, stereo를 넣어주었다.
그리고 add self loop 함수를 이용해서 자기 자신에게도 bond를 넣어주었다!
반응형
'연구' 카테고리의 다른 글
| 엔트로피 최대화 (0) | 2022.09.19 |
|---|---|
| mean field approximation 설명 (0) | 2022.09.18 |
| rdkit으로 SMILES를 canonical smiles로 바꾸는 법 (0) | 2022.09.14 |
| multiple sequence alignment(MSA) (0) | 2022.09.10 |
| empirical probability란 무엇일까 (0) | 2022.09.09 |